Pengambilan sampel formalin-fixed, paraffin-embedded (FFPE) merupakan metode yang banyak digunakan dan sederhana untuk menyimpan sample dalam penelitian kanker. Variasi struktural (SV) adalah proses mutasi utama dalam kanker, yang didefinisikan sebagai perubahan genom >50 bp dalam panjang, dan dapat memengaruhi gen atau kromosom secara keseluruhan. Pemeriksaan formalin dapat merusak dan mendegradasi molekul DNA namun, pengurutan sekuens DNA masih tetap mungkin dilakukan.
Dalam menggunakan teknologi sekuensing bacaan pendek tradisional, urutan panjang ini harus akan difragmentasi selama prosesnya sehingga kemungkinana kita kehilangan informasi struktur variasi genome cukup besar. Sehingga dapat menghasilkan identifikasi SV yang kurang tepat dan lengkap.
Oxford Nanopore sebagai long-read sequencing technology telah mengoptimalkan workflow yang dirancang khusus untuk mengekstraksi dan mensekuensing DNA sehingga dapat memberikan bacaan yang jauh lebih Panjang hingga beberapa kilobase dari sampel FFPE, memungkinkan deteksi dan analisis SV yang akurat. Dengan urutan sekuensing nanopore yang panjang, SV dapat dikarakterisasi secara komprehensif di seluruh genom kanker. Di sini, Oxford Nanopore Technology merancang workflow sederhana untuk analisis SV pada DNA yang diekstraksi dari sampel FFPE menggunakan PromethION™ Flow Cell.
Gambar 1
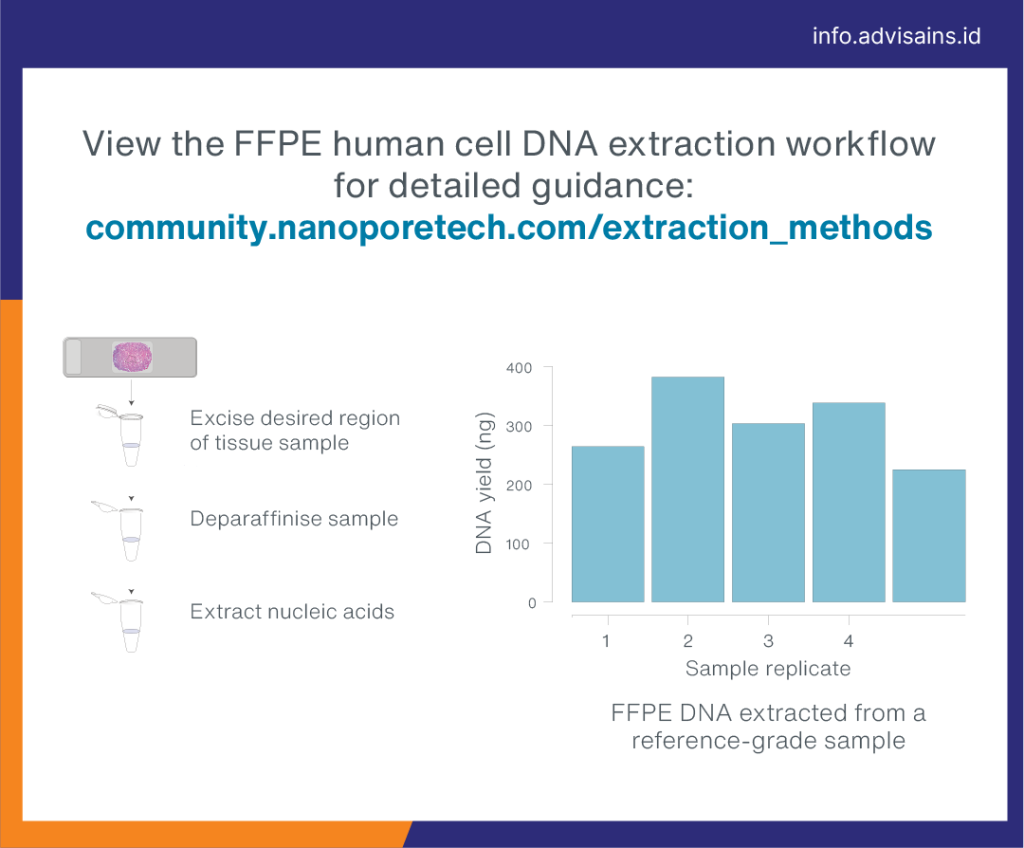
EKSTRAKSI: Mengisolasi, Mengukur, dan Menilai Integritas Sampel
Usia dan variasi persiapan sampel akan memengaruhi baik kualitas maupun tingkat fragmentasi DNA yang diekstraksi dari sampel FFPE. Alur kerja Oxford Nanopore ini dirancang dengan hati-hati untuk meminimalkan fragmentasi tambahan. Dimulai dengan penghilangan lapisan lilin parafin dari sampel, dilanjutkan dengan ekstraksi DNA menggunakan Kit Jaringan DNA FFPE dari Qiagen. Kami merekomendasikan untuk melakukan QC terhadap hasil ekstraksi DNA menggunakan instrumen Spectrofluorometer dan kualitas sampel melalui instrumen Nanospektrofotometer sebelum melanjutkan ke tahap library preparation.

Gambar 2
LIBRARY PREPARATION: Memilih kit sequencing yang tepat untuk sample
Tidak diperlukan fragmentasi DNA tambahan saat mempersiapkan gDNA yang diekstraksi dari FFPE untuk nanopore sequencing. Untuk memastikan molekul panjang yang mengandung informasi SV diperkaya, kami merekomendasikan untuk memilih fragmen DNA di atas ~1.5–2 kb menggunakan seleksi ukuran berbasis SPRI bead sebelum tahap library preparation. Kit Persiapan Perpustakaan Ligation (lihat gambar di bawah), pendekatan persiapan perpustakaan tanpa PCR, kemudian dapat digunakan untuk memaksimalkan hasil sekuensing. Namun, jika sampel FFPE sangat terdegradasi, hasil terbaik dapat diperoleh dengan mempersiapkan sampel menggunakan Kit Sekuensing PCR.
Gambar 3
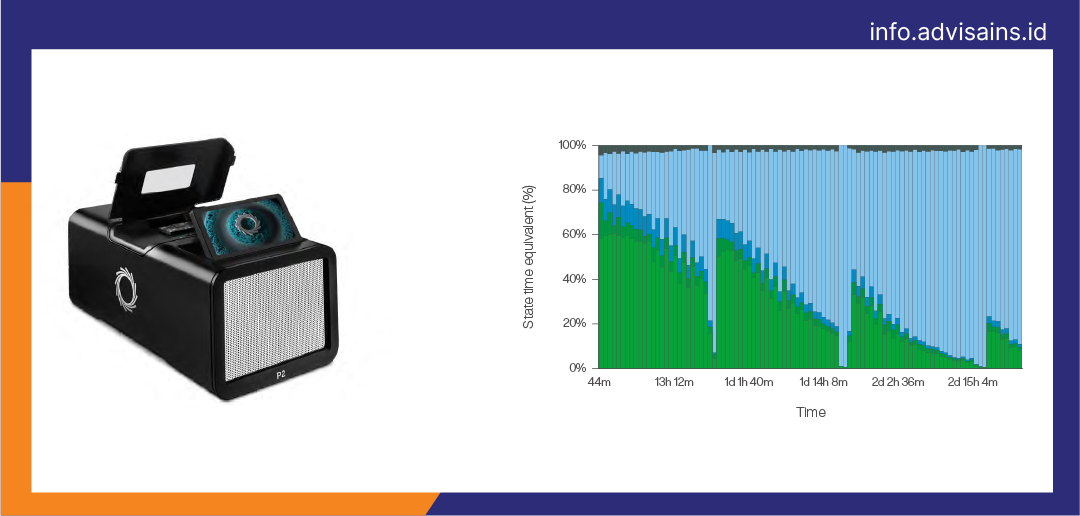
SEQUENCING: utilising high-output PromethION Flow Cell
Untuk metrik panggilan SV terbaik di seluruh genom manusia, kami merekomendasikan sekuensing hingga dengan kedalaman ≥30x (depth coverage), 15x depth coverage untuk whole genome sequencing. Kualitas sampel FFPE awal dapat memengaruhi hasil sekuensing, Jika anda mungkin ingin memeriksa kualitas perpustakaan Anda dengan sekuensing sejumlah kecil menggunakan Flongle Flow Cell atau minion flow cell menggunakna Minion atau GridION device untuk mengetahui secara real-time performa sequencingnya sebelum beralih ke sekuensing dengan kedalaman liputan yang lebih tinggi.
Untuk memaksimalkan output, kami merekomendasikan melakukan sekuensing pada PromethION flow cell. Seri perangkat PromethION menampilkan P24 dan P48 yang kuat dan dapat diletakkan di atas meja. Dikonfigurasi untuk sekuensing hingga 24 atau 48 PromethION flow cell, with high-performance integrated compute. Sementara, perangkat P2 yang kompak menyediakan fleksibilitas dua flow cell PromethION independen dan berkapasitas tinggi untuk kebutuhan sampel dengan throughput yang lebih rendah.
Gambar 4:
ANALYSIS: detecting SVs from FFPE sample sequencing data
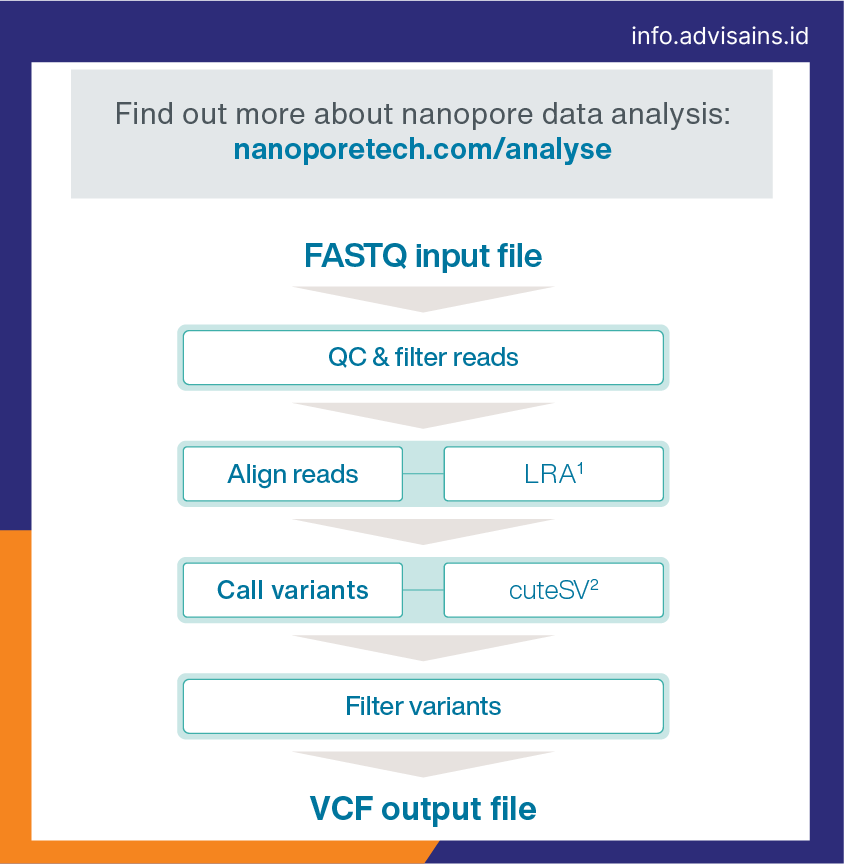
Untuk memanggil varian struktural (SV) germline dalam data nanopore sequencing Anda, kami merekomendasikan penggunaan alur kerja wf-human-sv. Tersedia di GitHub, alur kerja ini menggunakan file FASTQ yang dihasilkan selama sekuensing, melakukan penyelarasan dengan genom referensi FASTA menggunakan LRA1, dan memanggil penyisipan, penghapusan, dan duplikasi >50 bp menggunakan cuteSV2. Workflow ini ini menghasilkan file varian yang dipanggil (VCF) dan laporan QC.
Jika Anda mempelajari SV somatik dalam sampel penelitian kanker, kami merekomendasikan penggunaan alat Sniffles, menggunakan mode ‘somatik’. Untuk dataset sampel penelitian klinis tumor:normal berpasangan, varian somatik dapat dianalisis menggunakan nanomonsv.
view nanopore data analysis tools and pipelines on GitHub: github.com/nanoporetech
Advisains dapat memfasilitasi diskusi lebih lanjut mengenai product ONT. Sampaikan Kebutuhan Riset Anda Tim advisor spesialis kami sangat antusias mendukung keberhasilan riset dan aplikasi rutin Anda. Maka, jangan ragu untuk chat sekarang (+62 817 9154 607/info@advisains.id).
Baca Juga: Panduan Lengkap HPLC
Find out more at Cancer research and sequencing | Oxford Nanopore Technologies
References
- Chaisson, M. et al. GitHub: LRA [Online]. Available at: github.com/ChaissonLab/LRA [Accessed: 21 June 2022].
- Jiang, T. et al. Long-read-based human genomic structural variation detection with cuteSV. Genome Biol. 21:189 (2020).
- Smolka, M. et al. Comprehensive structural variant detection: from mosaic to population-level. bioRxiv. doi: https://doi.org/10.1101/2022.04.04.487055 (2022).
- Shiraishi, Y. et al. Precise characterization of somatic structural variations and mobile element insertions from paired long-read sequencing data with nanomonsv. bioRxiv. doi: https://doi.org/10.1101/2020.07.22.214262 (2021). Oxford Nanopore
- Original article could be found at FFPE_cancer_research_workflow.pdf (nanoporetech.com)